Aspectos relevantes sobre a P.1 e variantes
de preocupação (VOC)
O aparecimento de mutações é um evento natural e esperado dentro do processo evolutivo de qualquer vírus RNA. Essas mudanças genéticas acontecem à medida que o vírus faz novas cópias de si mesmo para se espalhar e prosperar. A maioria das mutações é irrelevante e algumas podem até ser prejudiciais à sobrevivência do vírus (pressão seletiva negativa), mas outras podem torná-lo mais infeccioso (pressão seletiva positiva) ou ameaçador para o hospedeiro (os seres humanos).
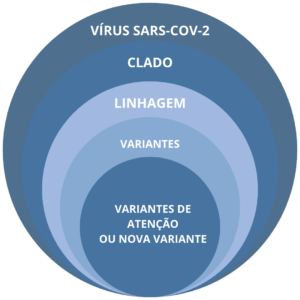
Baseado na evolução do SARS-CoV-2, pesquisadores desenvolveram um sistema de classificação em linhagens, tendo como base dois grandes ramos que receberam a denominação de A e B. Assim, à medida que foram surgindo diferenciações genéticas dentro de cada grande ramo, foram sendo designadas linhagens A.1, A.2, B.1, B.1.1, e assim sucessivamente. Desde a caracterização genômica inicial do SARS-CoV-2, este foi dividido em diferentes grupos genéticos ou clados. Quando ocorrem algumas mutações específicas, estas podem estabelecer uma nova linhagem do vírus em circulação. Em algumas referências, as linhagens são conhecidas como grupo genético. Também podem ocorrer algumas mutações adicionais e gerar diferenças dentro daquela linhagem. Quando isso acontece,
caracteriza-se uma nova variante e, quando as mutações ocasionam alterações relevantes clínico-epidemiológicas, como maiores gravidade e potencial de infectividade, essa variante é classificada como VOC, em inglês, variant of concern, em português traduzido para variante de atenção ou preocupação. A determinação de clado, linhagem, variante, entre outras, são utilizadas dentro de uma mesma espécie para melhor caracterizá-la e essas abordagens não correspondem a uma hierarquia taxonômica oficial. Didaticamente e, para fins de compreensão dos profissionais de vigilância em saúde, as classificações podem ser explicadas pela figura.
Mesmo que a maioria das mutações emergentes não tenha impacto significativo na disseminação do vírus, algumas mutações ou combinações de mutações podem fornecer ao vírus uma vantagem seletiva, como maior transmissibilidade ou capacidade de evadir da resposta imune do
hospedeiro. Estas variantes são consideradas preocupantes devido às mutações que podem conduzir ao aumento da transmissibilidade e ao agravamento da situação epidemiológica nas áreas onde forem identificadas. Desta forma, a vigilância de síndromes respiratórias, com atenção para a vigilância genômica, é importante para a saúde pública no enfrentamento da Covid-19.
NOVAS VARIANTES NO MUNDO
Existem três principais novas variantes sob a vigilância na corrida para compreender o que essas mutações podem impactar na situação epidemiológica no aumento da transmissibilidade.
- Variante VOC 202012/01, linhagem 1.1.7 – A nova variante VOC 202012/01 (Variante 01, ano 2020, mês 12), pertencente à linhagem B.1.1.7, foi notificada em dezembro/2020 pelas autoridades do Reino Unido à OMS. Esta variante contém 23 substituições de nucleotídeos e não está filogeneticamente relacionada ao vírus SARS-CoV-2 que circulava no Reino Unido no momento em que foi detectada. Essa linhagem carrega mutações definidoras, incluindo a mutação, N501Y na proteína S, que está associada a maior afinidade do vírus pelo receptor ACE-2 o que pode explicar a sua rápida expansão. O nome da mutação se refere à natureza da sua mudança: o aminoácido na posição 501 na proteína S mudou de N (asparagina) para Y (tirosina). De acordo com a OMS, a caracterização da VOC 202012/01 foi responsável por um aumento significativo da transmissibilidade, que contribuiu para aumentos na incidência e hospitalizações desde a segunda metade de dezembro/2020. Ainda não há evidências suficientes de que a VOC202012/01 esteja associada ao aumento de óbitos.
- Variante 501Y.V2, linhagem B.1.351 – A nova variante 501Y.V2 (Variante 02, ano 2020, mês 12), pertencente à linhagem 1.351, foi notificada em dezembro/2020 pelas autoridades da África do Sul à OMS. Esta variante é definida por várias alterações de proteína Spike (aminoácidos D80A, D215G, E484K, N501Y e A701V), e mais recentemente os vírus coletados têm alterações adicionais de aminoácidos L18F, R246I, K417N e deleção 242-244. Três das alterações (aminoácidos K417N, E484K e N501Y) estão localizadas no domínio de ligação ao receptor (RBD) da proteína Spike. Os resultados preliminares indicam que esta variante também pode sugerir um maior potencial de transmissibilidade. No entanto, ainda é necessária uma maior investigação sobre este e outros fatores que influenciam na transmissibilidade, severidade, imunidade, reinfecção, vacinação e diagnóstico.
- Variante P.1, linhagem B.1.1.28 – A nova variante VOC P.1 (Variante 03, ano 2021, mês 1), pertencente à linhagem B.1.1.28 (ou B.1.1.28.1) foi notificada em 09/01/2021, pela autoridade do Japão à OMS e ao Ponto Focal do Regulamento Sanitário Internacional (PFRSI) do A notificação descreveu uma nova variante em quatro viajantes provenientes de Manaus/AM. Esta variante apresenta mutações na proteína S (E484K, N501Y e K417Y), na região de ligação ao receptor, que geraram alterações de importância biológica e são semelhantes àquelas detectadas no Reino Unido e na África do Sul.
NOVA VARIANTE DO SARS-COV-2 NO BRASIL
Desde o ano 2000, como parte da rotina da vigilância dos vírus respiratórios, amostras coletadas são destinadas para sequenciamento genético. Com a pandemia da Covid-19, esses exames continuaram sendo realizados pelos Centros de Referência de Influenza no Brasil: Fundação Oswaldo Cruz, Instituto Adolfo Lutz e Instituto Evandro Chagas. Outros laboratórios públicos e privados, no Brasil, também realizam sequenciamento em suas linhas de pesquisa.
O sequenciamento genético não é um método de diagnóstico e não é realizado na rotina da confirmação laboratorial de casos suspeitos da Covid-19, tampouco é indicado para 100% dos casos positivos, contudo seu resultado permite quantificar e qualificar a diversidade genética viral circulante no país. Essa técnica exige investimentos em equipamentos, reagentes e recursos humanos em bioinformática e infraestrutura. Para a saúde pública, o sequenciamento genético do SARS-CoV-2, aliado a outros estudos, possibilita sugerir se as mutações identificadas podem influenciar na patogenicidade, transmissibilidade, além de direcionar medidas terapêuticas, diagnósticas ou contribuir no entendimento da resposta vacinal.
Por meio desse monitoramento, dados no site da Rede Genômica Fiocruz, hp://www.genomahcov.fiocruz.br/grafico/, até 28/01/2021, havia duas principais linhagens circulando no Brasil, desde fevereiro/2020: 35% B.1.1.33 e 32% B.1.1.28, ambas sem alterações significativas na proteína Spike. Porém, as consequências da manutenção de um cenário de ampla circulação e mobilidade humana na pandemia podem ser
observadas a partir de um novo protocolo de RT-PCR desenvolvido pela Fiocruz Amazonas, que demonstra uma dispersão geográfica de variantes de preocupação, com uma alta prevalência nas regiões avaliadas, como publicado em 04/03/2021. A ferramenta, capaz de detectar a mutação comum em três das variantes de preocupação (P.1, identificada no Amazonas, B.1.1.7, no Reino Unido e B.1.351, na África do Sul), foi utilizada nas unidades de apoio aos diagnósticos e centrais analíticas da Fiocruz para avaliação de cerca de mil amostras representativas de três regiões do Brasil (Sul, Sudeste e Nordeste). Dos oito estados avaliados neste primeiro recorte nacional apenas dois não tiveram prevalência da mutação associada às variantes de preocupação superior a 50%: Minas Gerais (30,3%) e Alagoas (42,6%). Embora o teste seja capaz de detectar uma mutação comum a três variantes de preocupação, há indicações de que a prevalência observada nos estados esteja associada à P.1, uma vez que as outras duas variantes não têm sido detectadas de forma expressiva no território brasileiro.
Estudos iniciais indicam que a variante descrita no Amazonas apresenta mutações (E484K e N501Y) que estão associadas à carga viral mais elevada e, consequentemente, maior capacidade de indivíduo portador transmitir o vírus. O ensaio de microneutralização demonstrou que as mutações E484K e N501Y podem causar baixa neutralização do patógeno pelos anticorpos, pois em mais de 50% das amostras de plasma convalescente, com anticorpos de expostos ao vírus, não obteve neutralização e nos 50% restantes obteve baixa neutralização, ou seja, quase metade dos indivíduos parecia que não ter sido infectada pelo microrganismo, sendo suscetíveis à nova infecção pelo SARS-CoV-2.
Nesse momento, é imprescindível entender o impacto dessa nova variante do SARS-CoV-2 na transmissão, gravidade da infecção, diagnósticos laboratoriais, tratamentos, vacinas ou medidas preventivas de saúde pública. A fim de limitar o impacto sobre as capacidades de detecção do vírus e suas variantes, a OMS recomenda o uso de ensaios diagnósticos para diferentes alvos (genes virais), o acompanhamento da transmissão local e atividades de prevenção e controle apropriadas às realidades de cada país. A maioria dos ensaios moleculares por PCR regularizados no Brasil utilizam mais de um alvo, o que reduziria o impacto ao diagnóstico, sendo importante este conhecimento.
- Edição 03. Março/2021. Assessoria Médica – Lab Rede
- Referências: 1. Brasil. NOTA TÉCNICA Nº 59/2021-CGPNI/DEIDT/SVS/MS.https://www.gov.br/saude/pt-br/media/pdf/2021/fevereiro/02-1/nota-tecnica-recomendacoes-quanto-a-nova-variante-do- sars-cov-2-no-brasi.pdf. 2. Brasil. NOTA TÉCNICA Nº 1/2021/SEI/GEVIT/GGTPS/DIRE3/ANVISA. https://www.gov.br/anvisa/pt-br/assuntos/paf/coronavirus/arquivos/arquivos- regulamentos/sei_anvisa-1285500-nota-tecnica.pdf 3. Freitas ARR, Giovanetti M & Alcantara LC, 2021. Interamerican Journal of Medicine and Health https://doi.org/10.31005/iajmh.v4i.181. 4. 4. Fiocruz https://portal.fiocruz.br/sites/portal.fiocruz.br/files/documentos/




Muito bem explicado.
Excelente